
发布时间: 2024-06-19 11:25:40
2024年6月14日,一篇题为Mendelian randomization supports causality between COVID-19 and glaucoma的孟德尔随机化研究论文发表于《Medicine (Baltimore)》,作者为中国学者。
这项研究采用双向双样本孟德尔随机化(MR)方法研究2019年冠状病毒病(COVID-19)与青光眼之间是否存在因果关系。研究揭示了肠道微生物群与淋巴瘤之间的实质性因果关系,该 MR 分析提供了 COVID-19 与青光眼之间潜在因果关系的初步证据。
1 摘要与主要研究
1.1 摘要
为了确定2019年冠状病毒病(COVID-19)与青光眼之间是否存在因果关系,采用2样本孟德尔随机化(MR)设计,采用逆方差加权的主要分析方法。采用异质性检验、多效性检验和留一法检验结果可靠性。根据 IEU 全基因组关联研究 (GWAS) 的数据,使用四组工具变量 (IV) 来调查 COVID-19 与青光眼风险之间的因果关系。结果显示,2组COVID-19(RELEASE)与青光眼风险显著相关[ID:EBI-a-GCST011071,OR(95%CI)=1.227(1.076-1.400),P=.002259;ID:EBI-a-GCST011073:OR (95% CI) = 1.164 (1.022–1.327),P = .022450;2 组 COVID-19 住院治疗与青光眼风险显著相关 (ID:ebi-a-GCST011081,OR (95% CI) = 1.156 (1.033–1.292),P = .011342;ID:EBI-a-GCST011082:OR (95% CI) = 1.097 (1.007–1.196),P = .034908)]。3种试验方法的灵敏度均为可接受(P > .05)。总之,该 MR 分析提供了 COVID-19 与青光眼之间潜在因果关系的初步证据。
1.2 SNPs和弱IV试验的结果
最后,我们从 IEU 中提取数据并分析了 2 组 COVID-19 相关 SNP(GWAS ID ebi-a-GCST011071 和 ebi-a-GCST011073)、2 组 COVID-19 住院相关 SNP(GWAS ID ebi-a-GCST011081 和 ebi-a-GCST011082)和 1 组青光眼(GWAS ID ebi-a-GCST009722)。来自 2 个 COVID-19 队列的 24 个 SNP 被用作遗传工具,2 个 COVID-19 住院队列与青光眼风险相关。所有设置为 IV 的 SNP 都具有 P < 5 × 10−8 和 F > 10。
1.3 两样本MR分析的结果
总体而言,使用不同方法进行 MR 估计后,COVID-19 与因青光眼住院的 COVID-19 之间存在显着的因果关联。结果显示COVID-19(EBI-a-GCST011071):OR(95%CI)=1.227(1.076~1.400),P=.002259;COVID-19(EBI-a-GCST011073):OR (95% CI) = 1.164 (1.022~1.327),P = .022450;COVID-19 住院(EBI-a-GCST011081):OR(95% CI)=1.156(1.033~1.292),P= .011342;COVID-19住院(EBI-a-GCST011082):OR(95% CI)= 1.097 (1.007~1.196),P = .034908。此外,MR-Egger 回归、加权中位数、简单模态和加权模态方法也产生了类似的结果。
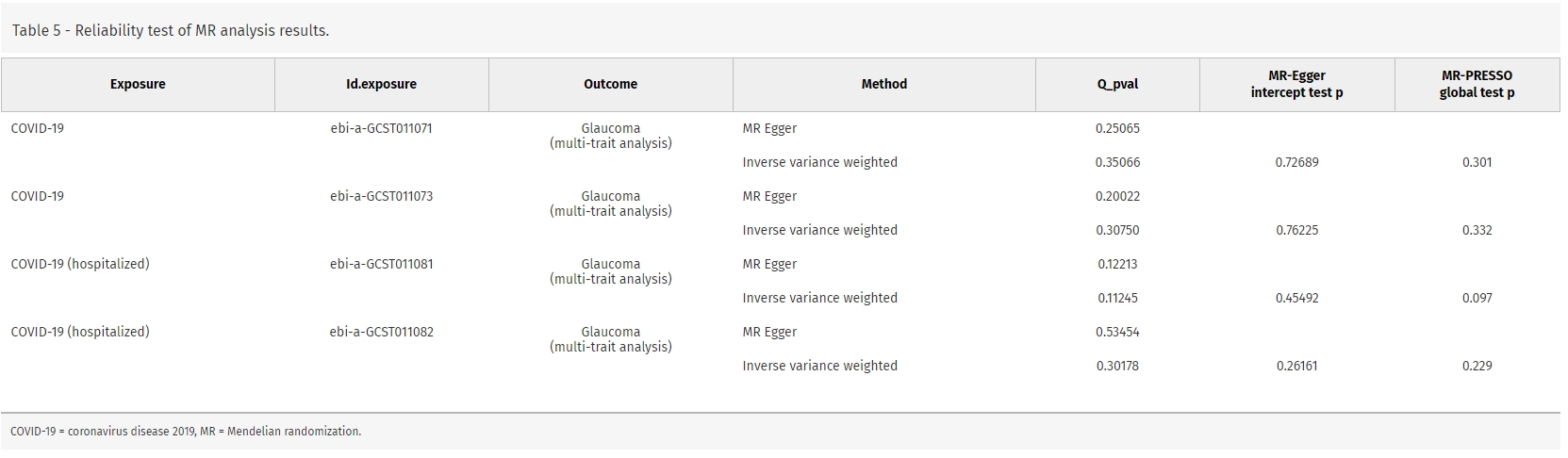
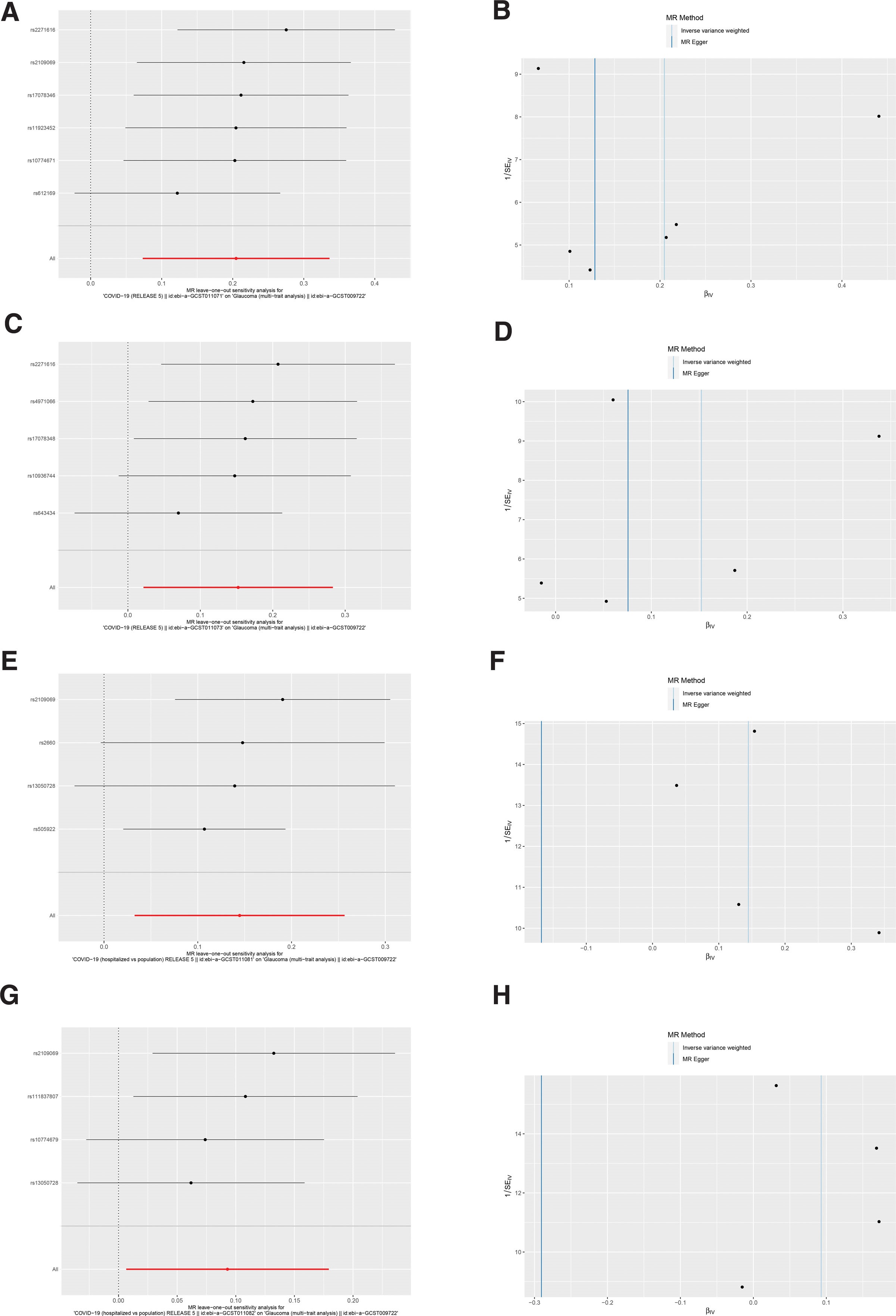
1.4 敏感性的评估
如表所示,MR-Egger截距和MR-PRESSO全局检验结果无统计学意义,表明MR分析结果可靠。留一法结果表明,逐渐去除各SNP后,剩余SNP的结果与原始结果相似,漏斗图大致呈对称性,表明在IV中未发现对结果有较强影响的SNP。
2 统计学方法
2.1 数据源
我们的数据均来自MRC综合流行病学部门(IEU)GWAS汇总日期(https://www.ebi.ac.uk/gwas/)。遗传仪器中与 COVID-19 相关的 SNP 基于对 COVID-19 和 COVID-19 住院的 GWAS 的 COVID-19 宿主遗传学倡议的研究,[27]大多数关于暴露的研究是在欧洲(55%)和美国(28%)进行的,英国(10%)和意大利(9%)。青光眼的结果[28]基因队列来自澳大利亚、英国和美国。总共招募了 4 组仪器变量来研究 COVID-19 在青光眼中的因果作用,包括与青光眼相关的 SNP(多性状分析),这些 SNP 是从 GWAS 的多性状分析中提取的。
2.2工具变量
为了获得合格的遗传变异作为工具变量 (IV),进行了一组质量控制分析。基于MR分析的3个核心假设,我们选择了与全基因组暴露独立相关的SNP(P < 5 × 10−8)作为仪器SNP。同时,我们设置参数(R2 <0.001 和 kb = 1000)以排除连锁不平衡,并丢弃与其他 IV 有更强相关性的变体。使用GWAS目录(https://www.ebi.ac.uk/gwas/)选择混杂因素。
2.3统计分析
使用 R 软件(版本 4.3.0,R 开发核心团队,奥地利维也纳)、TwoSampleMR(版本 0.5.7)和 MRPRESSO(版本 1.0)进行统计分析。我们使用 2 样本 MR 分析和反方差权重来评估 COVID-19 与青光眼之间的关系。Bonferroni 校正后 P 值< 5 × 10−8 被认为具有统计学意义。此外,还使用了其他方法来补充 MR 结果,包括 MR-Egger 回归、加权中位数、简单模态和加权模态方法。
采用异质性检验、多效性检验和留一法检测灵敏度。如果 MR-Egger 回归分析的多效性检验显示水平多效性,则应用 MR-PRESSO 异常值校正。
3 小结
这项孟德尔随机化(MR)研究使用双向双样本设计,探讨了2019年冠状病毒病(COVID-19)与青光眼之间的潜在因果关系。研究利用四组工具变量(IV)分析了COVID-19感染及住院治疗与青光眼风险的关联性,结果显示出显著的相关性。通过多种统计方法的验证,包括MR-Egger回归和加权中位数法,进一步强化了这一关联的可靠性。此外,灵敏度分析显示结果稳健,未发现强影响因素。总体而言,本研究提供了初步证据支持COVID-19可能与青光眼发病风险之间存在因果关系的观点,为进一步深入探讨二者关系奠定了基础。


下一篇:《JAMA-医学期刊上观察性研究干预效果的因果推断》文献翻译PART2