
发布时间: 2025-01-02 14:27:40
今天给大家带来的文章是发表在BioDataMin上的“Integrating transcriptomics and proteomics to analyze the immune microenvironment of cytomegalovirus associated ulcerative colitis and identify relevant biomarkers”。本研究的复杂之处在于有多个分组,在多个分组的情况下反复使用差异表达分析、WGCNA以及机器学习等来筛选生物标志物,最后构建疾病的诊断模型,虽然是普通的生信套路,但是也为具有多个分组的实验提供了参考。
题目:整合转录组学和蛋白质组学分析巨细胞病毒相关溃疡性结肠炎的免疫微环境并识别相关生物标志物
杂志:BioData Min
影响因子:IF=4.0
发表时间:2024.08.27
主要结果
1、CMV+IBD患者的免疫微环境与其他IBD患者不同
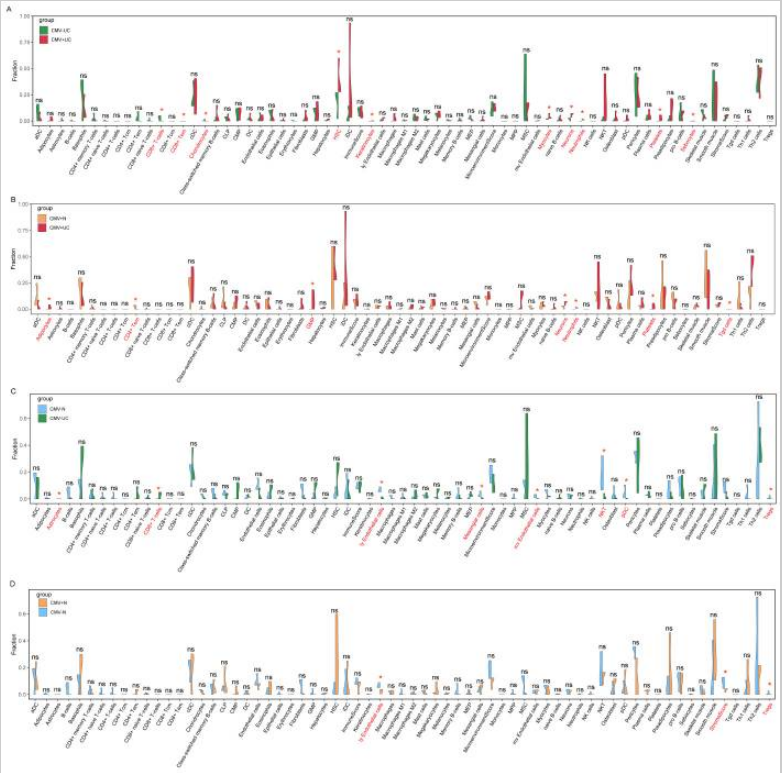
作者使用xCell算法评估了四个级别免疫细胞的丰度:CMV+UCvs.CMV-UC、CMV+UCvs.CMV+N、CMV-UCvs.CMV-N和CMV+Nvs.CMV-N。在CMV+UCvs.CMV-UC组中,发现CD8+T细胞、CD8+Tem、软骨细胞、HSC、角质形成细胞、肌细胞、神经元、中性粒细胞、血小板和皮脂细胞的丰度异常(图1A)。在CMV+UCvs.CMV+N组中,观察到脂肪细胞、CD4+Tem、GMP、神经元、中性粒细胞、血小板和Tgd细胞的水平异常(图1B)。在CMV-UCvs.CMV-N组中发现星形胶质细胞、CD8+T细胞、ly内皮细胞、系膜细胞、mv内皮细胞、NKT、pDC和Tregs的水平异常(图1C-D)。
2、识别CMV+UC特异性免疫微环境相关分子模块
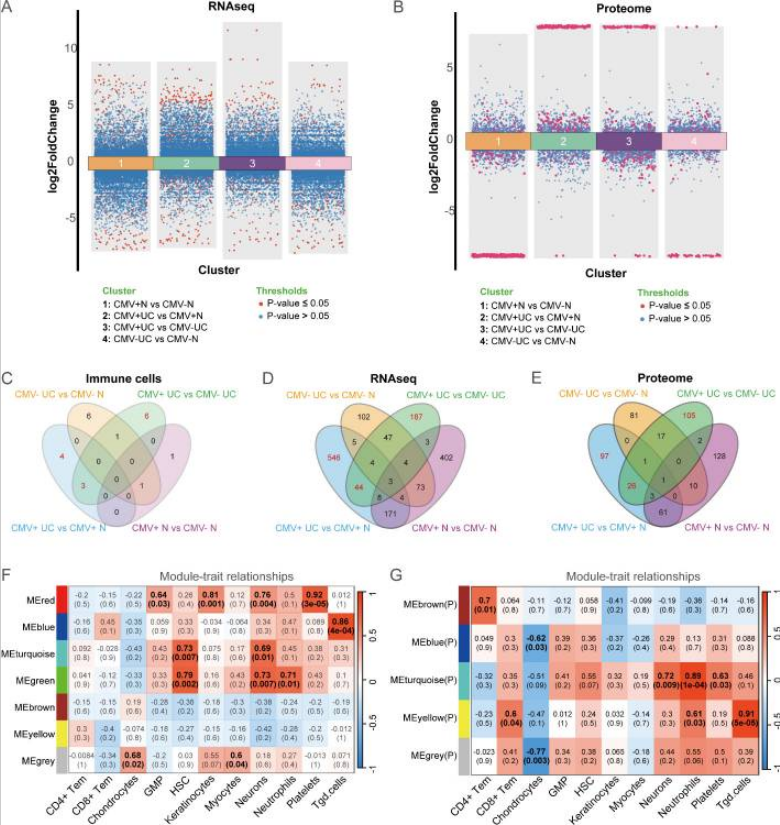
随后对这四组进行了转录和蛋白质水平的差异分析,筛选差异表达基因(图2A-B)以及通过GO和KEGG富集确定差异表达基因的功能。除此之外,还对四组中的异常免疫细胞、差异表达基因(DEG)和差异表达蛋白(DEP)进行了综合分析,以确定CMV+UC患者特有的免疫细胞和生物标志物(图2C-E)。接下来,使用WGCNA来识别CMV+UC患者特有的DEG和DEP的共表达模块及其与免疫细胞的关联(图2F-G)。
3、构建免疫细胞相关分子调控网络
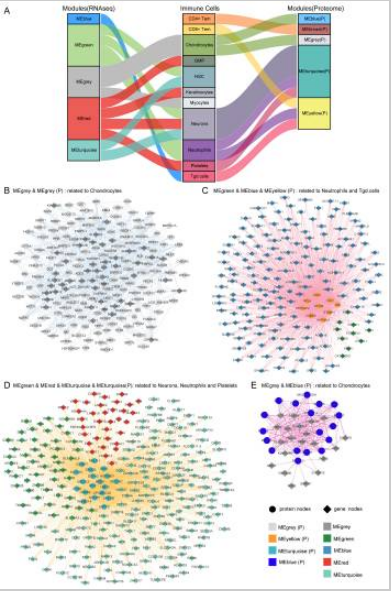
进一步整合了同一异常免疫细胞内的基因共表达模块和蛋白质共表达模块,确定了潜在的调控关系。分析表明,CD4+Tem细胞仅与MEbrown(P)模块相关;CD8+Tem细胞仅与MEyellow(P)模块相关等(图3A)。
进一步细化后,确定了四种潜在的调控关系:(I)MEgrey和MEgrey(P):与软骨细胞相关(图3B);(II)MEgreen、MEblue和MEyellow(P):与中性粒细胞和Tgd细胞相关(图3C);(III)MEgreen、MEred、MEturquoise和MEturquoise(P):与神经元、中性粒细胞和血小板相关(图3D);(IV)MEgrey和MEblue(P):与软骨细胞相关(图3E)。
随后,构建了不同调控关系的基因共表达模块和蛋白质共表达模块之间的共表达调控网络。只有绝对相关系数大于0.5且p值小于0.05的调控关系才被认为是显著的。这些调控网络可能反映了异常的免疫微环境受到多种因素的影响(图3B-E)。
4、使用机器学习算法识别CMV+UC的潜在诊断生物标志物
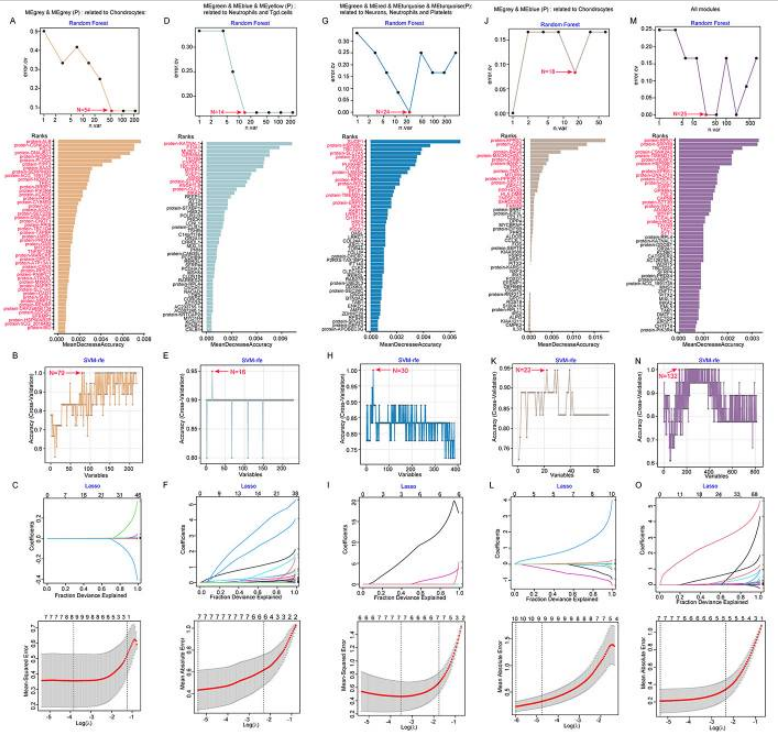
为了确定CMV+UC的早期诊断生物标志物,采用了五个层次的三种不同的机器学习算法:上面提到的四个调节网络和与异常免疫细胞相关的所有模块。
考虑到所有模块,随机森林算法确定了25个具有分类功效的基因/蛋白质(图4M),SVM-RFE算法选择了132个相关基因/蛋白质(图4N),LASSO算法确定了7个基因/蛋白质(图4O)
5、构建CMV+UC诊断模型
进一步整合了三种机器学习算法的结果。在所有模块中,我们鉴定出55种在三种算法中均能持续表现出有效分类的分子(图5A)。
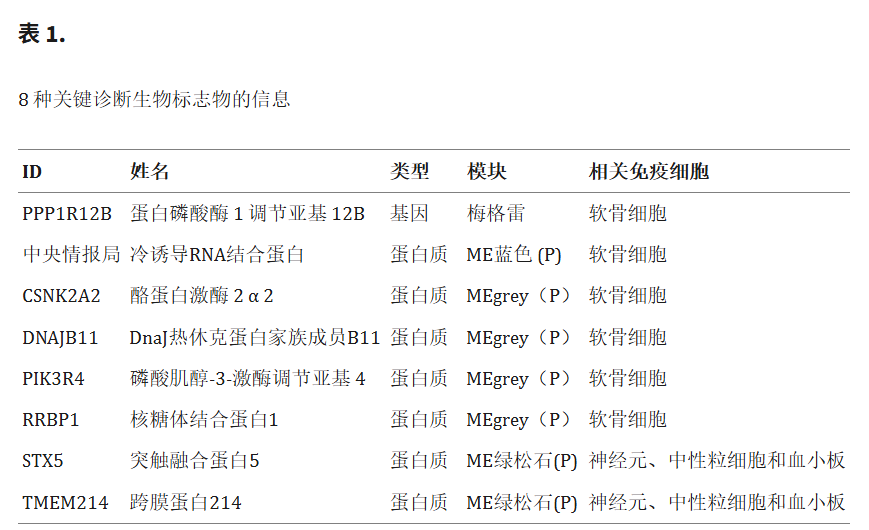
随后,整合了这些分子,得到了20个候选生物标志物,它们在不同水平上表现出稳定的分类性能。使用最优子集回归,构建了CMV+UC的诊断模型。分析显示,8个生物标志物模型实现了最大的R²和调整后的R²值,贝叶斯信息准则(BIC)最低(图5B)。这些结果表明,8个生物标志物模型(表1)在诊断CMV+UC方面表现最佳(图5C)。


上一篇:社会隔离与心绞痛风险的因果关联研究:基于孟德尔随机化的证据