
发布时间: 2024-12-12 10:16:33
小编今天分享的这篇文章是在Front Immunol上发表的题为“Identification of drug targets for Sjögren’s syndrome: multi-omics Mendelian randomization and colocalization analyses”的研究论文,通过研究DNA甲基化、基因表达和蛋白质数量性状位点与干燥综合征(SS)的关系,利用孟德尔随机化、共定位分析以及分子对接来识别SS的药物靶点。这篇文章抓住DNA甲基化和基因表达这一研究热点,对SS的治疗靶点进行了深入的分析,值得我们学习。另外数据是公开的,复现的可能性也非常高,换一个疾病也能做出SCI水平。
题目:干燥综合征药物靶点的识别:多组学孟德尔随机化和共定位分析
杂志:Front Immunol
影响因子:IF=5.7
发表时间:2024.06.12
研究背景
SS是一种难治性自身免疫性疾病,病理特征为进行性外分泌腺破坏,可累及口腔、眼、肾、肝、肺、关节、神经等多个器官。相较于其他自身免疫性疾病,SS与非霍奇金淋巴瘤的发病率显著升高有关,是与恶性肿瘤关系密切的疾病之一。临床治疗SS常用的润滑剂、糖皮质激素、免疫抑制剂等药物并非总是有效,且存在一定的不良反应,如局部过敏、胃肠道损害、皮肤损害等。因此,探索治疗SS的药物靶点具有深远的临床意义,可为研发治疗SS的新型药物提供理论支撑。通过遗传手段寻找药物靶点不仅可以大大提高药物研发的效率,还可以节省大量的人力和物力。此外,蛋白质作为分子通路的关键调控因子,已广泛成为药物靶点的主要来源。事实证明,由遗传关联支持的疾病相关蛋白质药物靶点获得上市批准的可能性更高。因此,基于遗传信息构建药物靶点是一种更有效的药物研发方法。
研究思路
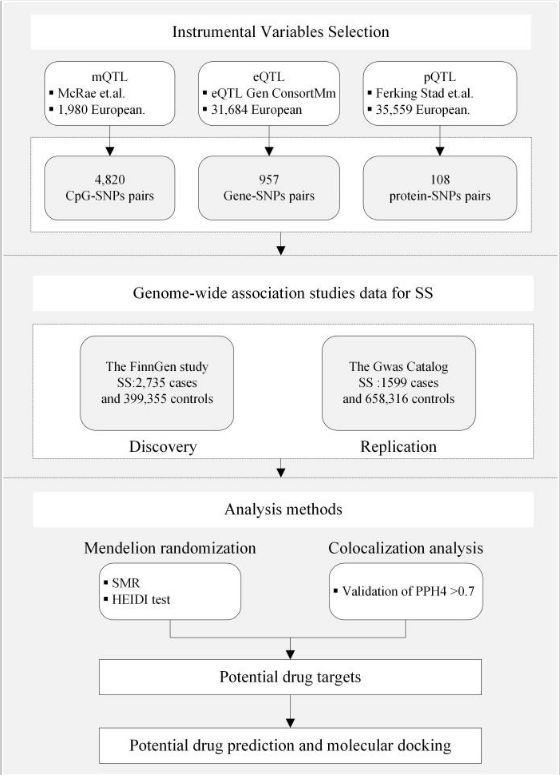
作者进行了基于汇总数据的孟德尔随机化(MR)分析,通过整合DNA甲基化、基因表达和蛋白质数量性状位点(分别为mQTL、eQTL和pQTL)来评估与SS相关的治疗靶点。与SS的遗传关联来自FinnGen研究(发现队列)和GWAS目录(复制队列)。共定位分析用于确定两个潜在相关表型是否在给定区域中共享相同的遗传因素。此外,为了更深入地探究DNA甲基化、基因表达和蛋白质丰度之间的潜在调控,我们进行了MR分析以探索候选基因甲基化与表达之间以及基因表达与蛋白质丰度之间的因果关系。进一步采用药物预测和分子对接来验证候选药物靶点的药理活性。
数据来源
甲基化数量性状基因座(mQTL)数据:
由McRae等人从1980年欧洲血统个体血液中的SNP-CpG关联中获得。
血液表达数量性状基因座(eQTL)数据:
来自eQTLGen联盟(https://eqtlgen.org/),包括31,684名个体、16,987个基因和31,684个来自血液样本的顺式eQTL。
蛋白质数量性状基因座(pQTL)数据:
来自一项针对35,559名冰岛人的大规模pQTL研究,提取了4907种循环蛋白质水平的遗传关联汇总统计数据。
SS数据源:
SS发现队列的GWAS数据来自FinnGenRelease10(https://www.finngen.fi/en),共包括2,735例SS病例和399,355例对照病例。验证队列来自GWAScatalog(https://www.ebi.ac.uk/gwas/),包括1,599例SS病例和658,316例对照病例(ID:GCST90018920)。
主要结果
DNA甲基化和SS
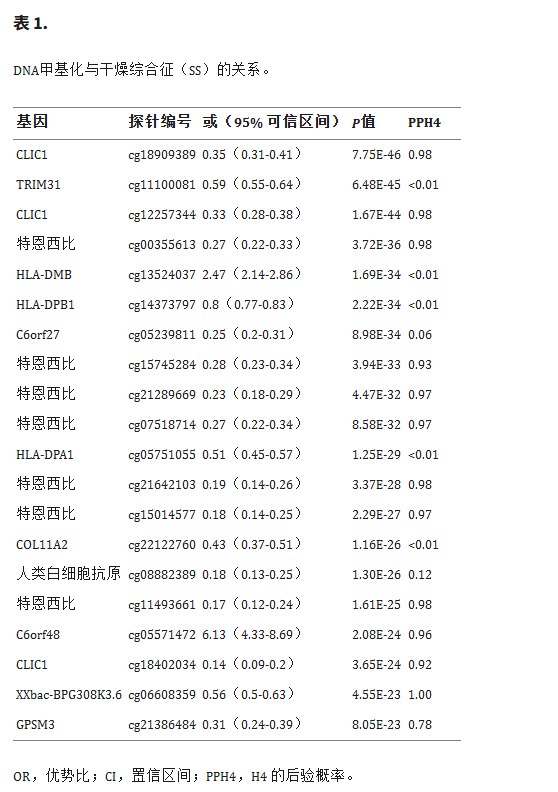
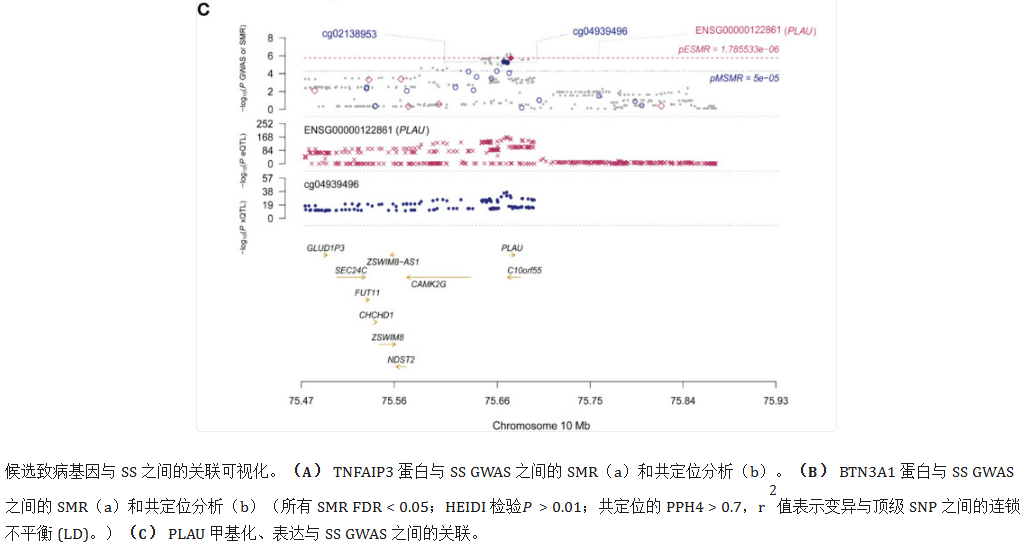
经过多重检验和共定位分析校正后,共鉴定出154个CpG位点与SS相关(P(FDR)<0.05,PPH4>0.70)(表1)。比如,位于CLIC1中的cg18909389和cg12257344,以及位于TNXB中的cg00355613、cg15745284、cg21289669和cg07518714与SS风险呈负相关。此外,位于C6orf48的cg05571472与SS风险呈正相关。在验证队列中,许多CpG位点(比如C6orf25(cg06132876)、PLAU(cg04939496)和TNXB(cg07237769))都得到了验证。
基因表达和SS
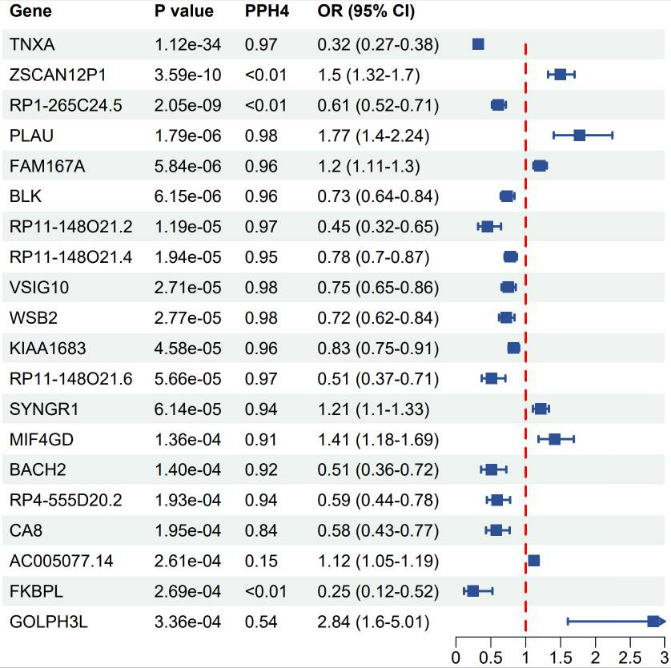
经过多重检验校正(P(FDR)<0.05)并进行共定位分析(PPH4>0.7)后,基因预测的CA8、BACH2、RP4–555D20.2、RP11–148O21.4、BLK、KIAA1683、RP11–148O21.2、TNXA、VSIG10和WSB2与SS风险呈负相关。相反,基因预测的PLAU、FAM167A、MIF4GD和SYNGR1与SS风险呈正相关(图3)。FAM167A、BLK、RP11–148O21.2、RP11–148O21.4、RP11–148O21.6、SYNGR1、MIF4GD和CA8与SS的关联在验证队列中得到了重现。
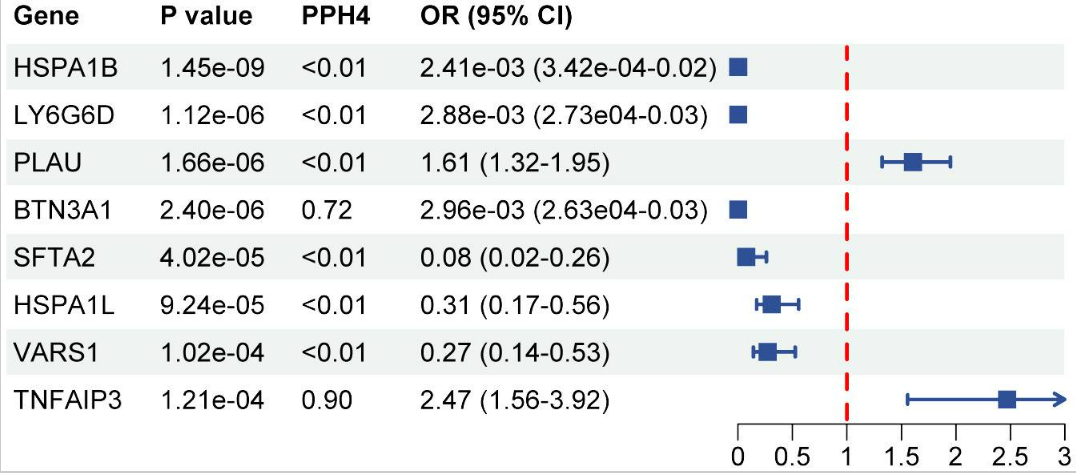
蛋白质和SS
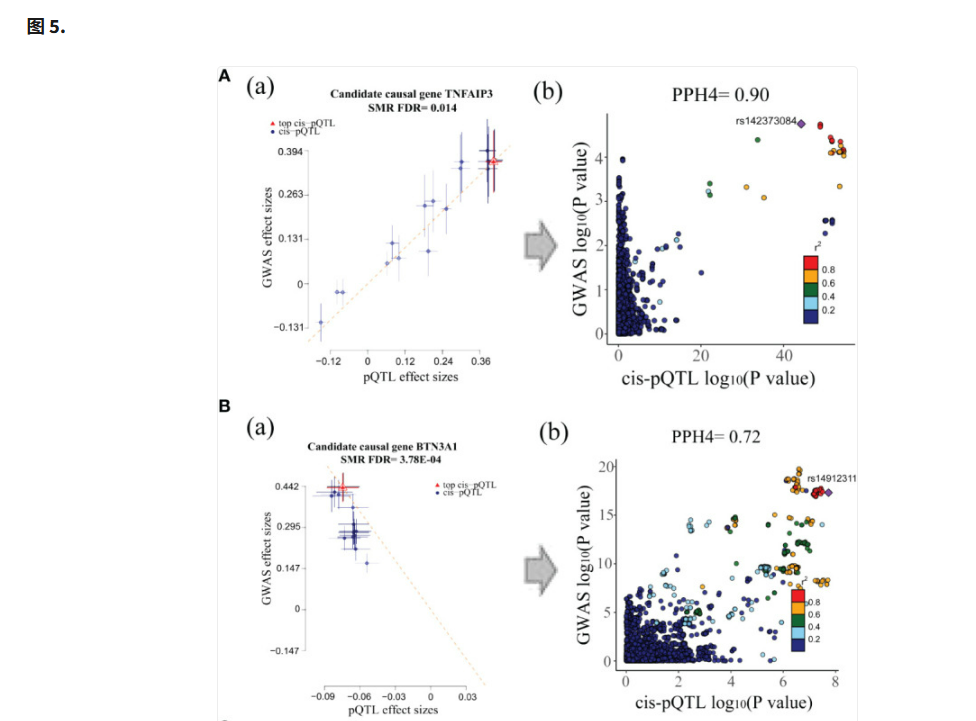
经过多重检验调整后,8种蛋白质与SS风险相关(P(FDR)<0.05)。HSPA1B、LY6G6D、BTN3A1、SFTA2、HSPA1L和VARS1与SS风险呈负相关。相反,PLAU和TNFAIP3与SS风险呈正相关(图4)。共定位分析的结果发现BTN3A1(PPH4=0.86)和TNFAIP3(PPH4=0.90)具有较高的共定位支持证据且BTN3A1在验证队列中得到了验证。
整合多组学层面的证据
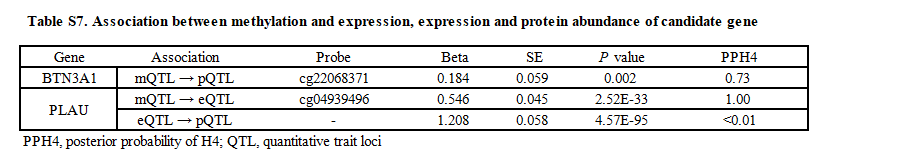
在整合多组学水平的证据后,确定了2个一级基因TNFAIP3和BTN3A1,以及二级基因PLAU(表2,图5)。在验证队列中,BTN3A1在循环蛋白水平上得到了验证(P(FDR)=0.036)。在探索基因甲基化、表达和蛋白质丰度之间的关联时,发现BTN3A1中cg22068371的甲基化与蛋白质水平呈正相关,这与cg22068371甲基化对SS风险的负面影响一致(补充表S7)。PLAU(cg04939496)的基因甲基化与基因表达之间以及基因表达与蛋白质水平之间也呈正相关,这与对SS风险的正相关相证实。在BTN3A1(cg22068371)的甲基化与蛋白质丰度之间以及PLAU(cg04939496)的基因甲基化与表达之间观察到强有力的共定位支持证据。
分子对接
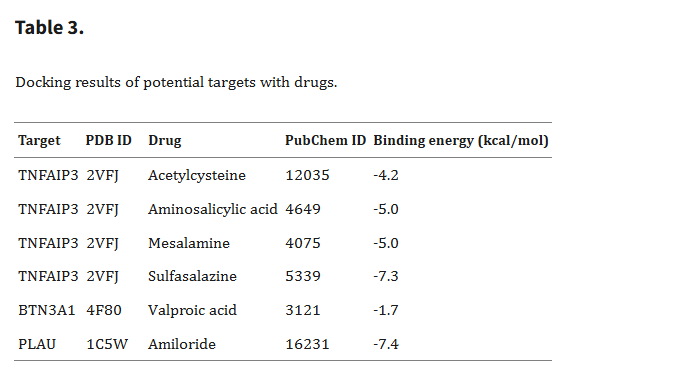
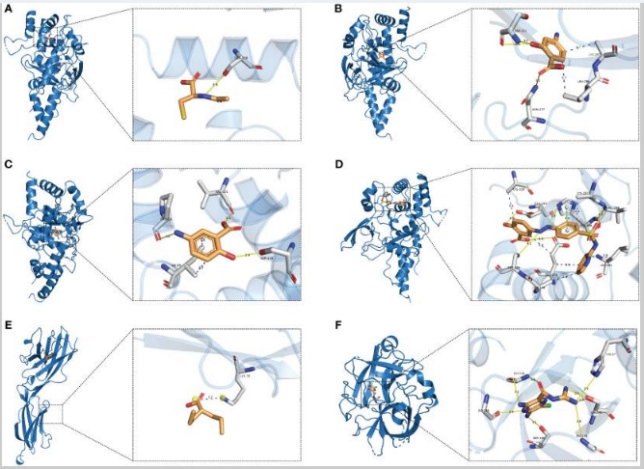
通过DrugBank查找到与靶标蛋白相关的候选药物,对应的药物ID和蛋白质结构数据见表3。PLAU-阿米洛利(-7.4kcal/mol)和TNFAIP3-柳氮磺胺吡啶(-7.3kcal/mol)具有最低结合能,被认为是配体和蛋白质之间最潜在的结合方式(图6)。
文章小结
研究通过整合DNA甲基化、基因表达和蛋白质丰度与SS的潜在因果关系,确定了TNFAIP3、BTN3A1和PLAU是SS的潜在靶点。这些发现为SS的靶向治疗提供了重要见解。
-END-

上一篇:解密循环酮体与肺癌及呼吸系统疾病的关联:基于孟德尔随机化的大规模GWAS分析
下一篇:孕期鱼类摄入与DHA补充——基于观察性研究的母婴健康影响分析